Bioconda生科根社区软件构建
发表于 2025/12/18
0
1. Bioconda及biconductor的介绍
1.1 Bioconda介绍
Bioconda 是一个专注于生物信息学软件的开源包管理系统,它基于Conda环境,为用户提供了一个便捷的方式来安装和管理各种生物信息学工具。Bioconda极大地简化了这些工具在不同操作系统(Linux和macOS)上的安装过程,解决了依赖关系问题,并确保了工具之间的兼容性。Bioconda社区维护着一个庞大的生物信息学软件库,这些软件被组织成不同的通道,用户可以通过简单的命令来安装所需的软件包。此外,Bioconda还鼓励贡献者提交新的或更新现有的软件包,以促进科学软件的可访问性和可持续性。
1.2 Bioconductor介绍
Bioconductor 是一个针对生物医学研究的开源软件项目,主要用于分析高通量基因组数据。该项目提供了一系列基于R语言开发的软件包、工作流以及注释数据集,旨在支持从原始数据处理到复杂统计分析等广泛的生物信息学任务。
Bioconductor最初于2001年发布,致力于与学术界紧密合作,推动基因组学数据分析方法的发展和传播。其资源广泛应用于基因表达分析、序列分析、基因组注释等领域,是生物信息学家和遗传学家的重要工具箱之一。
两者虽然服务于生物信息学的不同方面——Bioconda侧重于软件的获取和管理,而Bioconductor则更注重于提供具体的数据分析解决方案——但它们都极大促进了生物信息学领域的发展,帮助研究人员更高效地进行科学研究。
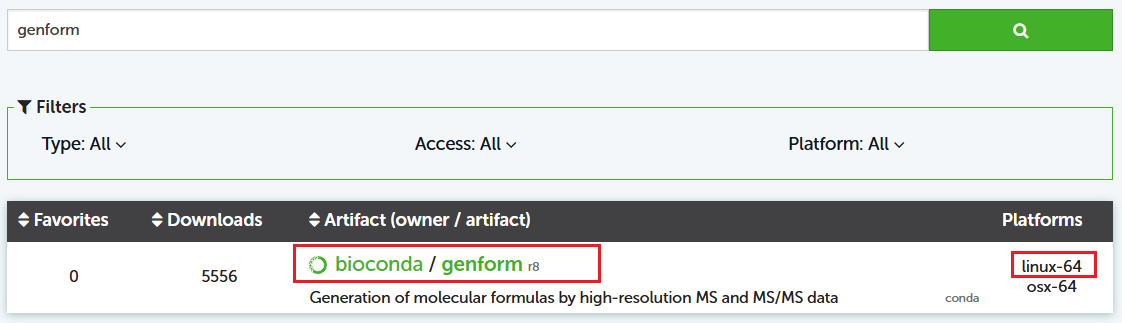
1.3 查看Bioconda社区软件适配清单
在社区搜索对应软件名称,以genform软件举例:
2. Anaconda的安装
2.1 Anaconda安装 (ARM)
1.获取Anaconda安装脚本
wget --user-agent="Mozilla/5.0 (Windows NT 10.0; WOW64) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/51.0.2704.103 Safari/537.36" https://mirrors.tuna.tsinghua.edu.cn/anaconda/archive/Anaconda3-2024.06-1-Linux-aarch64.sh2.执行安装脚本 , 注意:安装过程的交互要选yes
bash ./Anaconda3-2023.09-0-Linux-aarch64.sh3.加载环境
source ~/.bashrc4.验证,返回版本号即安装成功,不用执行第5步骤
(base) [root@ ~]# conda --version
conda 23.7.45.如果source ~/.bashrc执行失败,conda --version没有返回值可以尝试直接创建软连接
cd /usr/bin/
ln -s /root/anaconda3/bin/conda conda2. 2 环境配置
注意:必须执行, 否则conda build .等构建安装命令大概率失败
1.conda 添加channel, 使得在bioconda和conda-forge仓库的软件和本地构建的软件都能找到
conda config --add channels https://conda.anaconda.org/bioconda/linux-aarch64
conda config --add channels https://conda.anaconda.org/bioconda/noarch
conda config --add channels https://conda.anaconda.org/conda-forge/linux-aarch64
conda config --add channels https://conda.anaconda.org/conda-forge/noarch
conda config --add channels file:///root/anaconda3/conda-bld2.修改配置文件内容: vim ~/.condarc
channels:
- conda-forge
- bioconda
- defaults
- conda-bld
custom_channels:
conda-bld: file:///root/anaconda3
report_errors: true
channel_priority: strict3. Bioconda软件本地构建和测试
3.1 获取bioconda软件包资源
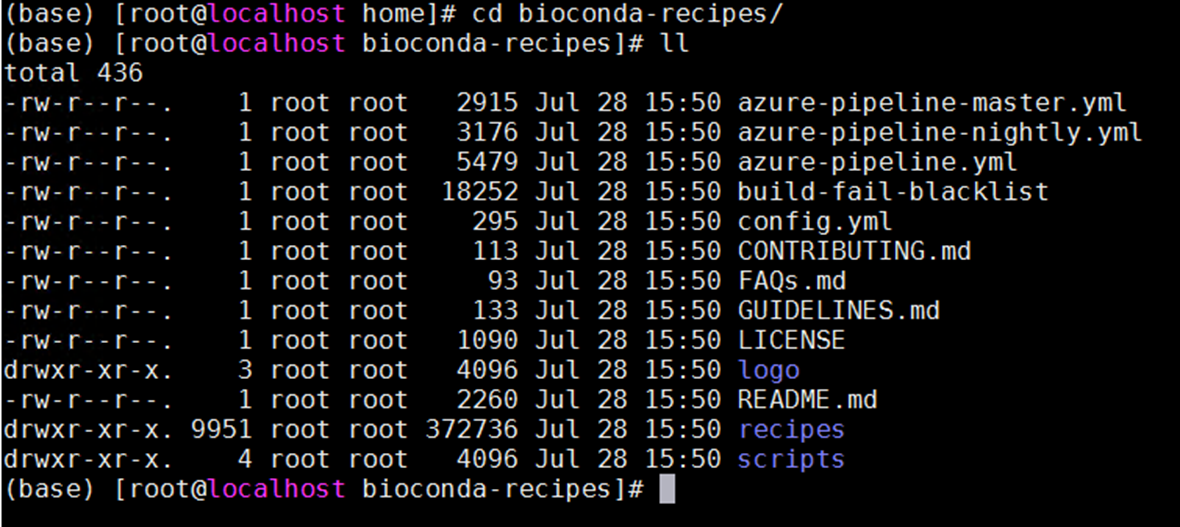
1. 先到github网站fork一下这个仓库: https://github.com/bioconda/bioconda-recipes
2. 然后在申请的服务器上执行git clone 你fork的仓库地址
3. clone后的目录下应该有以下内容, 其中需要适配的软件都在recipes目录下
3.2 本地构建软件
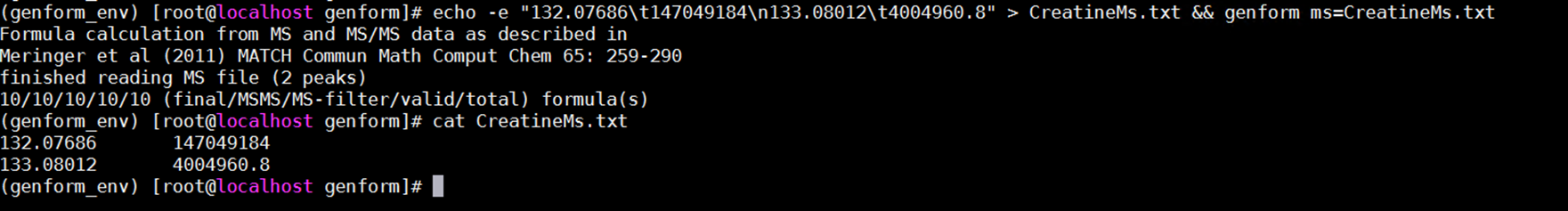
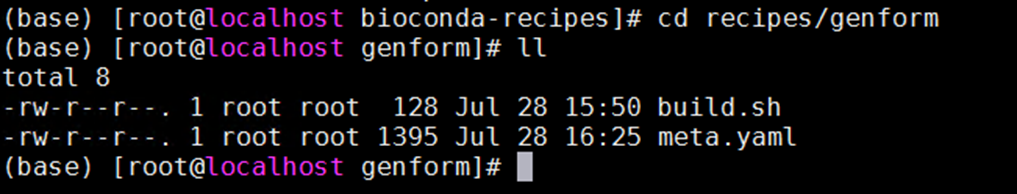
1.以 genform 软件适配为例(一般有一个构建脚本build.sh和一个配置文件meta.yaml)
2. meta.yaml 解析
package: #定义包的基本信息
name: genform
version: {{ version }}
source: #指定源代码位置
url:https://depot.galaxyproject.org/software/genform/genform_r8_src_all.zip
sha256: {{ sha256 }}
build: #定义构建时的相关信息
number: 8
skip: True # [osx]
run_exports: # run_exports这个要配置上, 不然提交可能check不通过
- {{ pin_subpackage(name, max_pin=None) }}
requirements: #定义依赖关系
build:
- {{ compiler('cxx') }}
test: #测试指令
commands:
- 'echo -e "132.07686\t147049184\n133.08012\t4004960.8" > CreatineMs.txt && genform ms=CreatineMs.txt'
about:
home: "https://sourceforge.net/projects/genform"
license: "GPL-2.0-only"
license_family: GPL
license_file: "src/lgpl-2.0.txt"
summary: "Generation of molecular formulas by high-resolution MS and MS/MS data.“
# 一般我们要加这个extra配置,指示aarch64架构也支持,但是如果在build下已经指示是noarch了,就不要加这个了
extra:
additional-platforms:
- linux-aarch643.获取meta.yaml文件
通常,我们在biconda-recipes仓库中,通过cd recipes/软件名等获取该软件资源,但有的软件可能已经被移出git仓库,这个时候不要慌。我们去这个官仓去搜一下,找到对应的软件下载bz2包,解压后可以得到。在recipes目录下,mkdir gclib && cd gclib, 然后将以上文件除conda_build_config.yaml和meta.yaml文件复制过来,执行mv meta.yaml.template meta.yaml,然后就可以按照正常的流程去适配了。
4. 本地构建
通过vim meta.yaml进行一系列操作后,就可以执行conda build .进行构建了。
# 一般有下面这个结果就表示build成功

如果构建成功则进行验证
通过 conda create –n genform_env genform –use-local –y 把刚刚构建好的包安装到新创建的环境genform_env中去。
执行conda activate genform_env 激活创建的环境
执行conda list | grep “genform”可以查看已经安装好的genform
conda create –n genform_env genform –use-local –y
conda activate genform_env
onda list | grep “genform”
5.本地测试
找到meta.yaml里test下的command复制命令,在当前安装了genform软件的环境下执行。