操作步骤
- 使用PuTTY工具,以root用户登录服务器。
- 执行以下命令添加gmap环境变量。
export PATH=/path/to/GMAP/Install/bin:$PATH
- 执行以下命令修改配置文件。
- 打开配置文件。
cp /path/to/PASA/PASApipeline-pasa-v2.4.1/pasa_conf/pasa.CONFIG.template /path/to/PASA/PASApipeline-pasa-v2.4.1/pasa_conf/conf.txt vim /path/to/PASA/PASApipeline-pasa-v2.4.1/pasa_conf/conf.txt
- 按“i”进入编辑模式,修改如下内容。
MYSQLSERVER=localhost MYSQL_RO_USER=test MYSQL_RO_PASSWORD=123456 MYSQL_RW_USER=pasa MYSQL_RW_PASSWORD=123456
- 修改完成后,按“Esc”键,输入:wq!,按“Enter”保存并退出编辑。
- 打开配置文件。
- 执行以下命令进入算例目录。
cd /path/to/PASA/PASApipeline-pasa-v2.4.1/sample_data
- 执行以下命令修改配置文件。
cp /path/to/PASA/PASApipeline-pasa-v2.4.1/pasa_conf/pasa.alignAssembly.Template.txt alignAssembly.config vi alignAssembly.config DATABASE=sample_data
修改完成后输入:wq!保存退出。
- 执行以下命令运行用例。
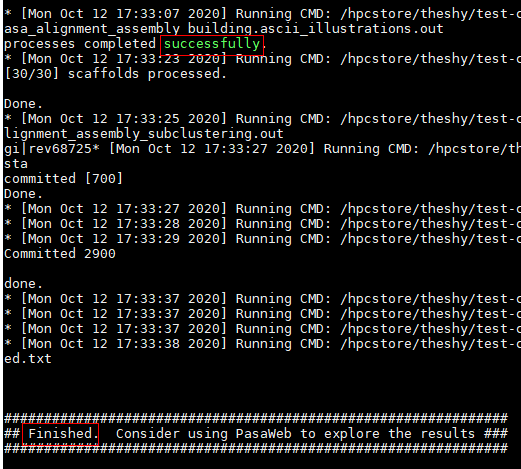
gzip -d genome_sample.fasta.gz { time Launch_PASA_pipeline.pl -c alignAssembly.config -C -R -g genome_sample.fasta -t all_transcripts.fasta.clean -T -u all_transcripts.fasta -f FL_accs.txt --ALIGNERS blat,gmap --CPU 96; } 2>&1 |tee PASA.log需要查看PASA.log日志,日志中打印Finished表示PASA正常结束。
输出的结果样例如图1所示。