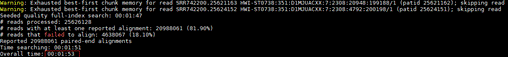运行和验证
操作步骤
- 使用PuTTY工具,以root用户登录服务器。
- 执行以下命令进入“man_hg19”目录。
cd /path/to/CHROMFA/man_hg19/
- 执行以下命令解压算例文件。
gzip -d SRR742200_1.fastq.gz gzip -d SRR742200_2.fastq.gz
- 执行以下命令运行Bowtie获取“.sam”文件(需要对应的生物序列文本“xxx.fastq”)。
bowtie -S -t -q -p 128 man_index -1 ./SRR742200_1.fastq -2 ./SRR742200_2.fastq SRR742200_out.sam
结果样例如下图所示。