操作步骤
- 使用PuTTY工具,以root用户登录服务器。
- 执行以下命令进入“/path/to/CASE”目录。
cd /path/to/CASE
- 执行以下命令设置HISAT执行文件的环境变量。
export PATH=/path/to/HISAT2/hisat2-2.1.0/:$PATH
- 执行以下命令解压表2中的“bdgp6.tar.gz”。
cd /path/to/CASE tar -xvf bdgp6.tar.gz cd bdgp6
- 执行以下命令修改“make_bdgp6.sh”文件。
- 打开“make_bdgp6.sh”文件。
vi make_bdgp6.sh
- 按“i”进入编辑模式,注释如下内容。
# ENSEMBL_BASE=ftp://xxxxxxxx
- 按“Esc”键,输入:wq!,按“Enter”保存并退出编辑。
- 打开“make_bdgp6.sh”文件。
- 使用STFP工具,将四个算例文件“Drosophila_melanogaster.BDGP6.dna.toplevel.fa.gz”、“Homo_sapiens.GRCh38.86.chr.gtf.gz”、“R1.fq.gz”和“R2.fq.gz”拷贝到“/path/to/CASE/bdgp6”目录下。
- 执行以下命令解压文件。
gzip -d Homo_sapiens.GRCh38.86.chr.gtf.gz gzip -d Drosophila_melanogaster.BDGP6.dna.toplevel.fa.gz gzip -d R1.fq.gz gzip -d R2.fq.gz
- 执行以下命令,重命名“fa”文件。
mv Drosophila_melanogaster.BDGP6.dna.toplevel.fa genome.fa
- 执行以下命令运行“make_bdgp6.sh”文件。
./make_bdgp6.sh
- 执行以下命令建立转录组。
extract_exons.py Homo_sapiens.GRCh38.86.chr.gtf > genome.exon extract_splice_sites.py Homo_sapiens.GRCh38.86.chr.gtf > genome.ss
- 执行以下命令将基因组和转录组建立索引。
hisat2-build -p96 genome.fa --ss genome.ss --exon genome.exon genome_tran
- 执行以下命令运行HISAT2。
hisat2 -t -p 96 -x genome_tran -1 R1.fq -2 R2.fq -S Test.sam
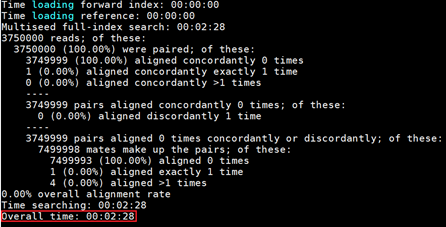
需要查看日志中“Overall time”数值,单位是“XX时:XX分:XX秒”,数值越小性能越优。
测试结果样例如图1所示。